ACHIEVEMENTS
![]()
ACHIEVEMENTS研究成果
-

- ACHIEVEMENTS
- タンパク質の抗体ラベリング技術を改良し、構造解析をアシスト 〜電子顕微鏡やX線結晶解析による構造決定を加速化〜
プレスリリース
2021.04.20
タンパク質の抗体ラベリング技術を改良し、構造解析をアシスト 〜電子顕微鏡やX線結晶解析による構造決定を加速化〜
横浜市立大学 大学院生命医科学研究科の禾 晃和准教授らは、筑波大学、大阪大学蛋白質研究所、京都大学、東北大学との共同研究で、タンパク質に外来の抗原配列を移植して抗体を結合させる技術を開発しました。本技術によって、これまで直接結合する抗体がなかったタンパク質に抗体を結合させることが可能になり、X線結晶解析[*1]や電子顕微鏡単粒子解析[*2]で立体構造情報が明らかになる可能性があります。
本研究成果は、科学誌「Acta Crystallographica Section D, Structural Biology」に掲載されました。(英国夏時間 2021年4月19日午前9時掲載)
- 【研究成果のポイント】
- ◆標的タンパク質の立体構造を壊さずに抗原配列を移植し、抗体を結合させてラベリングできる技術を開発した
- ◆抗体ラベリング[*3]の適用拡大で、電子顕微鏡やX線結晶解析による構造決定の可能性も広がった
研究背景
タンパク質の立体構造解析は、生命現象の解明だけでなく、創薬においても非常に重要な研究手法です。その解析において、抗体は有用な実験ツールとして用いられてきました。特にタンパク質の抗体によるラベリングは、X線結晶解析では、結晶になりにくいタンパク質の結晶化を促進させる効果があり、電子顕微鏡解析では、コントラストが低い画像から標的タンパク質を見つけ出す目印として役立ちます。しかしながら、抗体でラベリングを行うには、標的タンパク質を直接認識する抗体があることが前提で、適用範囲は限られていました。
この課題に対し、禾准教授らは新たな抗体ラベリング技術の開発に取り組んできました。これまでに、「PAタグ」と呼ばれる12残基のアミノ酸配列を標的タンパク質に移植し、このPAタグと強固に結合するNZ-1抗体でラベリングする技術を開発しました(図1)。このPAタグの移植部位を最適化することで、標的タンパク質にNZ-1抗体が安定に結合することが示されましたが、PAタグの移植やそのNZ-1抗体との結合によって標的タンパク質の一部の構造が変化してしまうことも明らかになっていました。
研究内容
そこで今回の研究では、タグの長さを伸ばすことで移植した際の標的タンパク質の構造変化を抑えることを試みました。その結果、PAタグのN末端側にアミノ酸残基を2つ付け加えたPA14タグでは、標的タンパク質を天然の構造に近い状態に維持できることが示されました。
標的タンパク質にPA14タグを移植してX線結晶解析を行ったところ(図2)、PA14タグはNZ-1抗体と結合すると末端同士が近づいてリング状の構造をとること(図3)、そしてNZ-1抗体が結合しても標的タンパク質の立体構造がほとんど壊れないことが確かめられました(図4)。さらに分子動力学シミュレーション[*4]からも、PA14タグを移植した標的タンパク質の構造が安定に維持されることを確認しました。そして、PA14タグを細胞膜の中で働くタンパク質に移植し、NZ-1抗体を結合させて負染色電子顕微鏡解析[*5]を行うことで、標的タンパク質の構造情報を取得することにも成功しました(図5)。
今後の展開
今回の研究から、PA14タグは標的タンパク質に移植して抗体を結合させるのに適した配列であることが確かめられましたが、その一方で、標的タンパク質に結合したNZ-1抗体の向きが完全には固定されず、揺らぐ場合があることも分かりました。今後、標的タンパク質の構造は壊さずに抗体の向きを固定できるようになれば、クライオ電子顕微鏡などを用いて精密に立体構造を調べることが可能になると期待されます。
論文情報
タイトル:Moving toward generalizable NZ-1 labeling for 3D structure determination with optimized epitope tag insertion
著者:Risako Tamura-Sakaguchi, Rie Aruga, Mika Hirose, Toru Ekimoto, Takuya Miyake, Yohei Hizukuri, Rika Oi, Mika K. Kaneko, Yukinari Kato, Yoshinori Akiyama, Mitsunori Ikeguchi, Kenji Iwasaki, Terukazu Nogi
掲載雑誌:Acta Crystallographica Section D, Structural Biology
DOI:https://doi.org/10.1107/S2059798321002527
研究費
※本研究は、日本学術振興会科研費、国立研究開発法人日本医療研究開発機構(AMED)創薬等ライフサイエンス研究支援基盤事業(BINDS)、横浜市立大学 学術的研究推進事業 研究奨励プロジェクトの助成を受けて実施されました。
用語説明
*1 X線結晶解析:
結晶化した物質にX線を照射して回折パターンを解析し、立体構造情報を取得する研究手法。タンパク質のような巨大な分子でも結晶化すれば、解析が可能になる。
*2 電子顕微鏡単粒子解析:
タンパク質試料に電子線を照射して撮影した透過像から立体構造情報を取得する技術。重金属塩を満たしてタンパク質に浸潤させ形状を解析する負染色電子顕微鏡法[*5]とタンパク質を薄い氷の中に閉じ込めて構造を解析するクライオ電子顕微鏡法が代表的な解析手法である。
*3 抗体ラベリング:
標的となるタンパク質に抗体を結合させる技術。立体構造解析で利用する際は、抗原結合部位を含む抗体の一部分を結合させる場合が多い。
*4 分子動力学シミュレーション:
計算機シミュレーションの1つで、分子を構成する各原子の運動を解析する研究手法。実験的に観測が難しい、分子の動的な性質を解析することが可能となる。
*5 負染色電子顕微鏡解析:
タンパク質などの生体試料を重金属塩で染色すると、試料の隙間や周囲に重金属塩が浸潤する。染色後の試料に電子線を照射すると、主に電子散乱能の大きい重金属塩が形成するコントラストにより間接的にタンパク質など生体試料の形状を観察できる。透過像で実際に観察しているのは重金属塩の分布であることから負染色と呼ばれる。
図、画像、表
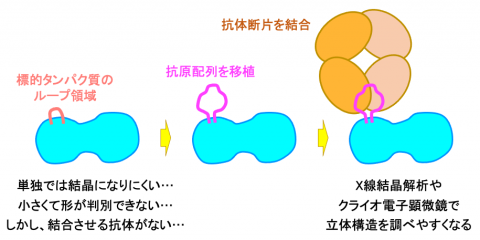
図1 外来の抗原配列を利用した抗体ラベリング法
標的タンパク質のループ領域に、外来の抗原配列を移植し、これを認識する抗体の断片を結合させることで、特異的に結合する抗体がない標的タンパク質にも抗体ラベリング法を用いることが可能になる。
抗体ラベリング法は、X線結晶解析では結晶化を促進させる効果があり、クライオ電子顕微鏡でも小さくて判別が難しいタンパク質を画像の中から見つけ出すのに役立つことから、立体構造解析での有用なツールとなっている。
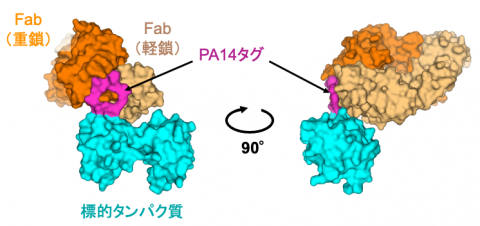
図2 PA14タグの移植によって作製した抗体断片と標的タンパク質の複合体
水溶性のタンパク質を標的タンパク質として用いて、PA14タグの挿入と抗体断片の結合が可能かどうかを調べた。水色の標的タンパク質の表面からマジェンタ色のPA14タグが突き出し、濃淡2色のオレンジ色で示した抗体のFab断片が結合している様子が、X線結晶解析で明らかになった。
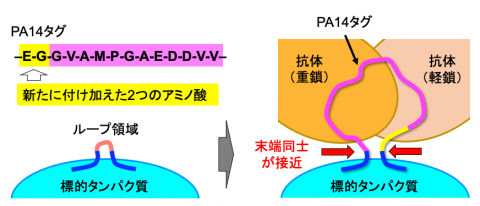
図3 PA14タグの配列と抗体との結合状態
PA14タグは、12個のアミノ酸からなるPA12タグの先頭にE(グルタミン酸)とG(グリシン)の2つのアミノ酸を付け加えたものである。新たに付け加えた2つのアミノ酸によって、抗体と結合した時のPA14タグは、最初と最後のアミノ酸が接近することが可能になり、全体では閉じたリング状の形をとる。このような性質があることで、分子表面から飛び出していないループ領域にもPA14タグを移植して抗体を結合させられることが、今回の研究でも確かめられた。
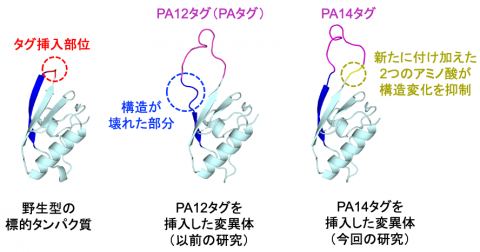
図4 タグの移植と抗体の結合によって生じる標的タンパク質の構造変化
以前の研究と今回の研究では、いずれも標的タンパク質のβ-ヘアピン領域の先端に抗原配列を移植した(左の図)。以前の研究で、PA12タグ(一般的には、PAタグと呼ぶ)を移植して抗体を結合させた時は、青色で示したβ-ストランド構造が壊れてしまう問題が起きた(真ん中の図)。今回、黄色の2つのアミノ酸を付け加えたPA14タグを移植して抗体断片を結合させたところ、青色のβ-ストランド構造は、タグを挿入していない野生型とよく似た状態に保たれることがわかった(右の図)。
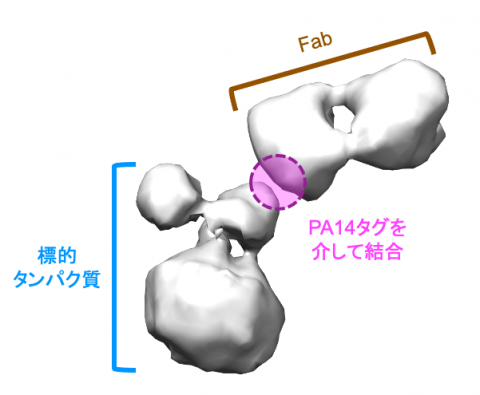
図5 PA14タグによる抗体ラベリング法の電子顕微鏡解析での応用
PA14タグを膜タンパク質(膜に埋もれた状態ではたらくタンパク質)に移植し、抗体ラベリングを行った。この膜タンパク質の水溶性の領域にあるループにPA14タグを移植した変異体を細胞の中で発現させてから、界面活性剤で膜から溶かし出し、抗体断片を結合させた。この膜タンパク質と抗体断片の複合体に重金属溶液を添加し、負染色電子顕微鏡単粒子解析によって概形を調べた。抗体断片や膜タンパク質の水溶性ドメインの形状がはっきりとわかるデータが得られた。
研究体制
横浜市立大学 大学院生命医科学研究科
准教授 禾 晃和
教授 池口 満徳
助教 浴本 亨
筑波大学生存ダイナミクス研究センター
教授 岩崎 憲治
大阪大学 蛋白質研究所
特任研究員(常勤) 廣瀬 未果
京都大学 ウイルス・再生医科学研究所
教授 秋山 芳展
助教 檜作 洋平
東北大学未来科学技術共同研究センター / 大学院医学系研究科
教授 加藤 幸成
参考文献
PAタグを利用した抗体ラベリング技術の開発の先行研究に関する論文
タイトル:Application of the NZ‐1 Fab as a crystallization chaperone for PA tag‐inserted target proteinsz
著者:Risako Tamura, Rika Oi, Satoko Akashi, Mika K. Kaneko, Yukinari Kato, Terukazu Nogi
掲載雑誌:Protein Science (2019) 28, 823-836
DOI:10.1002/pro.3580






